Программное обеспечение для составления отчетности virSEAKдля импортацииcеквенирование нового поколения (NGS)для молекулярной биологии
Добавить в папку «Избранное»
Добавить к сравнению
fo_shop_gate_exact_title
Характеристики
- Функция
- для составления отчетности, для импортации, cеквенирование нового поколения (NGS)
- Применение
- для молекулярной биологии
- Тип
- автоматизированное
Описание
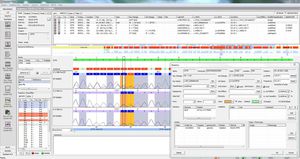
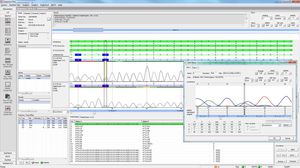
virSEAK (RUO - research use only) предлагает быстрое и удобное сравнение вашей последовательности SARS-CoV-2 с имеющимися последовательностями. После индивидуальной настройки вашего набора просто загрузите данные fastq- или fasta и получите выровненную последовательность с вариантами.
Кроме того, ваша последовательность будет отнесена к роду панголинов (например, B.1.1.7) и кладу GISAID (например, L).
virSEAK также автоматически проверяет, содержит ли ваша последовательность варианты белка шипа N501Y и или E484K.
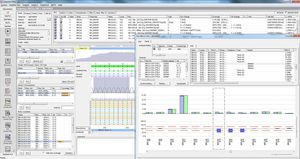
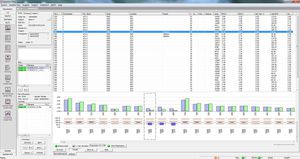
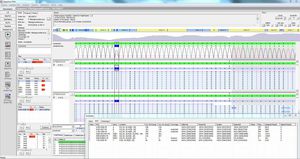
Показатели покрытия и качества, включая: требуемое, абсолютное, среднее и медианное покрытие, % оснований "дикого типа" и "N". Индикатор качества показывает, удовлетворяет ли качество определенным порогам качества.
Кроме того, инструмент оптимизирован для высокопроизводительного секвенирования NGS с автоматизированными функциями импорта и экспорта и настраиваемым переносом последовательности. Легко создавать мультифаста-файлы для улучшения отчетности в центральный офис по наблюдению за эпидемиологической ситуацией и распространением мутаций (например, для Германии - Институт Роберта Коха).
virSEAK - устанавливаемая версия:
простой импорт сырых fastq или предварительно обработанных fasta файлов секвенирования
высокопроизводительное секвенирование NGS с автоматическим пакетным импортом и экспортом
автоматическое оповещение при наличии мутации (мутаций) N501Y и/или E484K
получение отнесения к роду панголинов и кладу GISAID
проверка наличия других вариантов с указанием частот
сравнить подтипы, количество и расположение находок, ...
экспортировать последовательность и варианты (с метаинформацией, в формате fasta и/или csv)
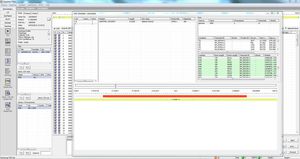
хранение результатов
полностью автоматизированное высокопроизводительное секвенирование с настраиваемой передачей последовательности
---
Каталоги
Для этого товара не доступен ни один каталог.
Посмотреть все каталоги JSI medical systemsРасширенный поиск
- Программное обеспечение для анализа
- Программное обеспечение для составления отчетности
- Автоматизированное программное обеспечение
- Модуль программного обеспечения
- Программное обеспечение для импортации
- Программное обеспечение сервер
- Модуль программного обеспечения для анализа
- Программное обеспечение для молекулярной биологии
- Модуль программного обеспечения для лабораторий
- Модуль программного обеспечения для захвата
- Модуль программного обеспечения для контроля
- Программное обеспечение cеквенирование нового поколения (NGS)
- Модуль программного обеспечения для картографии
* Цены указаны без учета налогов, без стоимости доставки, без учета таможенных пошлин и не включают в себя дополнительные расходы, связанные с установкой или вводом в эксплуатацию. Цены являются ориентировочными и могут меняться в зависимости от страны, цен на сырьевые товары и валютных курсов.