- Продукция
- Каталоги
Программное обеспечение для секвенирования нового поколения GensearchNGSдля анализадля управления даннымидля диагностики
Добавить в папку «Избранное»
Добавить к сравнению
fo_shop_gate_exact_title
Характеристики
- Функция
- для анализа, для управления данными, для диагностики
- Применение
- для секвенирования нового поколения, для лабораторий
Описание
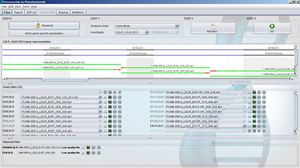
GensearchNGS - это интегрированное программное решение для анализа данных ДНК-Seq, полученных с помощью широко используемого оборудования NGS, такого как Roche/454, Illumina, Ion Torrent и Proton. Он был спроектирован и разработан в тесном сотрудничестве с ведущими аккредитованными группами по диагностике в Европе и при поддержке проекта NMDchip FP7.
Он объединяет все этапы от импорта и фильтрации последовательности считывания, разделения штрих-кода, выравнивания, определения вариантов и примечаний к окончательному отчету об обнаруженных вариантах. Варианты могут быть аннотированы различными общественными и частными источниками данных (Ensembl, Alamut, Genome Trax,...) Они могут быть отфильтрованы с помощью логических комбинаций между выборками для семейных исследований.
Основные характеристики:
Удобное в использовании программное обеспечение для повторного опроса проектов NGS в диагностической среде, работающее на системах Windows, Linux и MacOS.
Подключается*) к различным инструментам интерпретации и базам данных, например: Ensembl, dbSNP, Alamut, Clinvar, Wiki Pathways, Gene Ontology*).
Фильтруйте варианты по образцам (например, варианты, общие для нескольких образцов или исключающие варианты одного образца из другого).
Система базы данных, ориентированная на пациента, для управления данными последовательности и валидированными вариантами.
Возможна персонализация для экспорта данных в вашу систему управления лабораторией.
Возможность публикации ваших вариантов на Clinvar*).
Разработано совместно с аккредитованными лабораториями.
Для более крупных проектов (WES/WGS): возможность параллельного использования нескольких ПК.
Технология плагина: выравнивание с нашими запатентованными или общедоступными алгоритмами, такими как Bowtie, BWA или Stampy.
Аннотация и фильтрация с помощью дескрипторов и кодов HPO, OMIM и Orphanet, полезны при поиске мутаций в целых экзомах или целых геномах.
---
Расширенный поиск
- Программное обеспечение для анализа
- Программное обеспечение для лабораторий
- Программное обеспечение для диагностики
- Программное обеспечение для управления данными
- Программное обеспечение для молекулярной биологии
- Программное обеспечение для генетики
- Программное обеспечение для секвенирования нового поколения
* Цены указаны без учета налогов, без стоимости доставки, без учета таможенных пошлин и не включают в себя дополнительные расходы, связанные с установкой или вводом в эксплуатацию. Цены являются ориентировочными и могут меняться в зависимости от страны, цен на сырьевые товары и валютных курсов.